ศูนย์จีโนมทางการแพทย์ รพ.รามาธิบดี เผยผู้เชี่ยวชาญทั่วโลกประสานความร่วมมือเฝ้าติดตาม “FU.1” หรือ XBB.1.16.1.1 หลานของ XBB.1.16 มีความได้เปรียบในการเติบโต-แพร่ระบาดสูงกว่า XBB.1.16 ถึง 50% ไทยพบแล้ว 1 คน
ตระกูลโอมิครอนกลายพันธุ์ 3 กลุ่มหลัก
เมื่อวันที่ 17 พ.ค. ศูนย์จีโนมทางการแพทย์ คณะแพทยศาสตร์ โรงพยาบาลรามาธิบดี มหาวิทยาลัยมหิดล เผยข้อมูลผ่านเฟซบุ๊ก Center for Medical Genomics ระบุข้อมูลสถานการณ์สายพันธุ์โควิด-19 ว่า แม้จำนวนผู้ติดเชื้อโควิด-19 ทั่วโลกจะลดลงอย่างมีนัยสำคัญ แต่โควิด-19ตระกูลโอมิครอนยังมีการกลายพันธุ์แพร่ระบาดอย่างต่อเนื่อง ที่เด่นชัดมี 3 กลุ่ม คือ
1.XBB.1.5 นามแฝงคือ คราเคน (Kraken) 2.XBB.1.16 นามแฝงคือ อาร์คทูรัส (Arcturus) และ 3.XBB.1.9.1 นามแฝงคือ ไฮเปอเรี่ยน (Hyperion)
ไทยเจอ 2กลุ่ม XBB.1.16 และ XBB.1.9.1
“กลุ่มโอมิครอน XBB.1.5 มีการลดจำนวนลงอย่างต่อเนื่อง ประเมินว่าจะถูกแทนที่โดย กลุ่ม XBB.1.16 และ XBB.1.9.1 ที่พบระบาดมากที่สุดในอินเดีย และมีการกระจายไปทั่วโลก ส่วนในอาเซียนพบในสิงคโปร์และไทย”
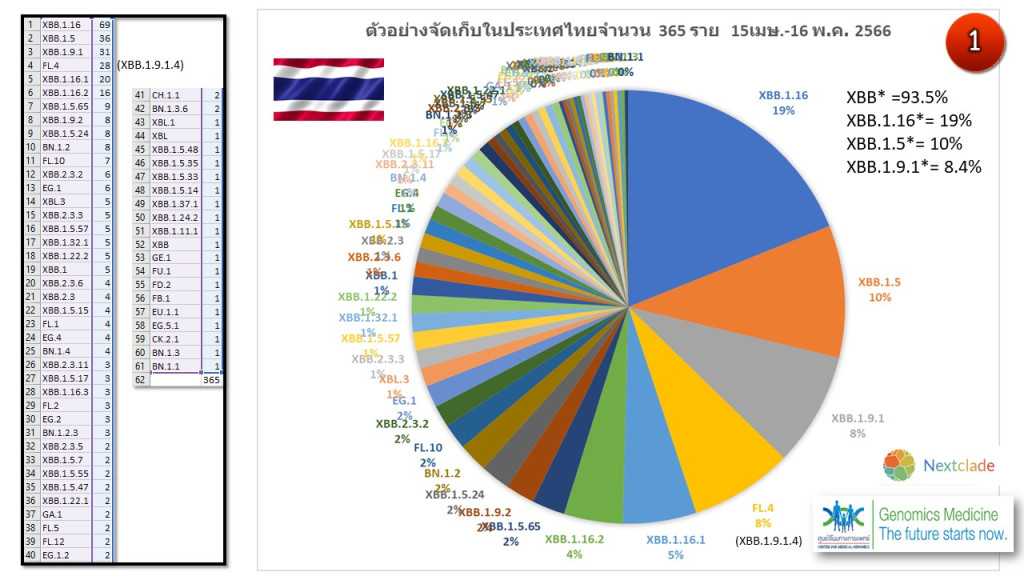
ศูนย์จีโนมฯ พบไทยเจอสายพันธ์ย่อย XBB.1.16 ประมาณ 19%
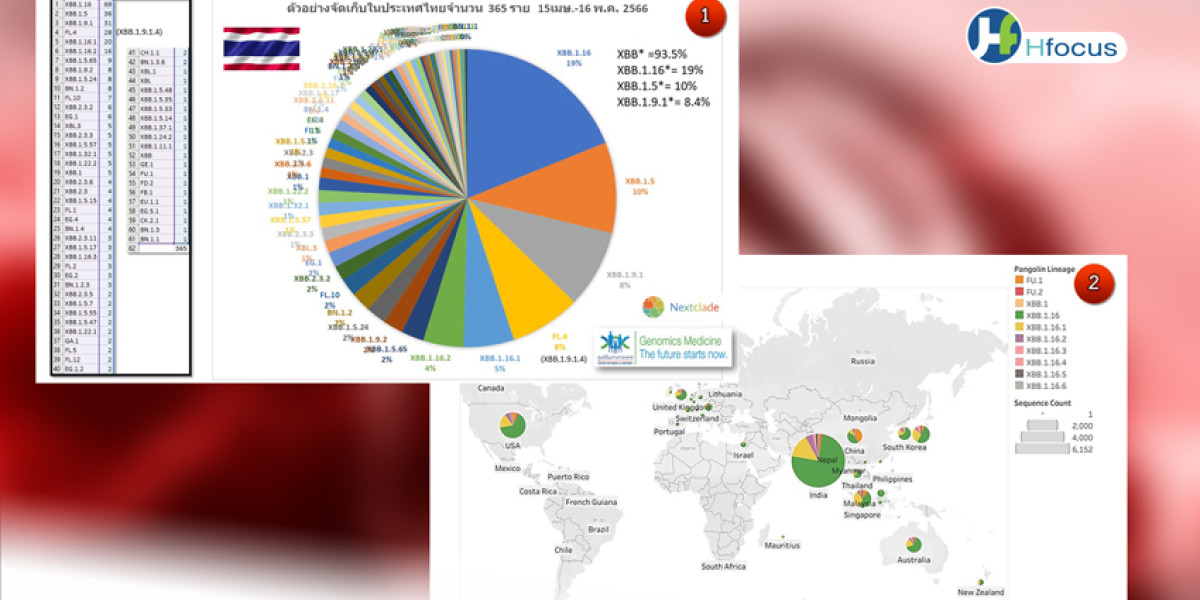
ศูนย์จีโนมทางการแพทย์ รพ. รามาธิบดีติดตามการกลายพันธุ์ในระดับจีโนมของไวรัสโคโรนา 2019 ทั้งในประเทศไทยและทั่วโลกจากการถอดรหัสพันธุกรรมทั้งจีโนมของไวรัสโควิด-19 โดยการเก็บตัวอย่างจากผู้ติดเชื้อในประเทศและข้อมูลรหัสพันธุกรรมจากฐานข้อมูลโควิดโลก (GISAID) ในช่วง 30 วันที่ผ่านมา ระหว่างวันที่ 15 เม.ย. -15พ.ค. 2566 พบโอมิครอนสายพันธุ์หลักในประเทศไทยเป็นกลุ่ม XBB ประมาณ 93.5% ประกอบด้วยสายพันธุ์ย่อยอันดับหนึ่งคือ XBB.1.16 ประมาณ 19% อันดับสองเป็น XBB.1.5 ประมาณ 10% และอันดับสามเป็น XBB.1.9.1 ประมาณ 8.4%
สายพันธุ์ย่อย XBB.1.16 พบกลายพันธุ์ระบาดทั่วโลก 3 รุ่น
สายพันธุ์ย่อยอันดับหนึ่งกลุ่ม XBB.1.16* หรืออาร์คทูรัส (Arcturus) พบการกลายพันธุ์ระบาดไปทั่วโลกถึง 3 รุ่นคือ
รุ่นแรก
โอมิครอน XBB.1.16 (กลายพันธุ์ ณ. ตำแหน่ง S:E180V, S:478R) ทั่วโลกพบ 9,003 ราย ประเทศไทย 139 ราย
รุ่นลูก
โอมิครอน XBB.1.16.1 (S:T547I) ทั่วโลกพบ 2,714 ราย ประเทศไทย 26 ราย
โอมิครอน XBB.1.16.2 (ORF3a:V13L, ORF1a:P926H) ทั่วโลกพบ 666 ราย ประเทศไทยพบ 25 ราย
โอมิครอน XBB.1.16.3 (A2893C) ทั่วโลกพบ 175 ราย ประเทศไทยพบ 5 ราย
โอมิครอน XBB.1.16.4 (S:T678I) ทั่วโลกพบ 177 ราย ประเทศไทยยังไม่พบ
โอมิครอน XBB.1.16.5 (T9991C,C16332T) ทั่วโลกพบ 135 ราย ประเทศไทยยังไม่พบ
โอมิครอน XBB.1.16.6 (S:F456L) ทั่วโลกพบ 23 ราย ประเทศไทยยังไม่พบ
รุ่นหลาน
โอมิครอน XBB.1.16.1.1 (T3802C)**: นามแฝง FU.1 ทั่วโลกพบ 122 ราย ประเทศไทยพบ 1 ราย
โอมิครอน XBB.1.16.1.2 (C8692T): นามแฝง FU.2 ทั่วโลกพบ 149 ราย ประเทศไทยยังไม่พบ
ทั่วโลกติดตามสายพันธุ์ FU.1 หรือ XBB.1.16.1.1
สายพันธุ์ย่อยในกลุ่มโอมิครอน XBB.1.16* โดยเฉพาะ “FU.1 (XBB.1.16.1.1)” ถือได้ว่าเป็น “รุ่นหลาน” ของ XBB.1.16 พบการระบาดมากในนครเซี่ยงไฮ้ ประเทศจีน ผู้เชี่ยวชาญทั่วโลกได้ประสานความร่วมมือให้ช่วยกันเฝ้าติดตามเนื่องจากมีค่าความได้เปรียบในการเติบโต-แพร่ระบาด (relative growth advantage) สูงกว่า XBB.1.16 ถึง 50%
ในขณะที่กลุ่มโอมิครอน XBB.1.16* “รุ่นลูก” คือ “XBB.1.16.1” พบการแพร่ระบาดในสวีเดน โดยมีโอมิครอนกลุ่ม “XBB.1.9.1” และรุ่นลูกและรุ่นหลาน “FL*” กลายพันธุ์แพร่ระบาดตามมาติดๆ คาดว่าอาจเข้ามาแทนที่กลุ่ม XBB.1.16 ในอนาคต
ศูนย์จีโนมฯรามา เผยเหตุผลดำเนินการถอดรหัสพันธุกรรมโควิด
ศูนย์จีโนมทางการแพทย์ รพ. รามาธิบดีดำเนินการถอดรหัสพันธุกรรมโควิด-19 ทั้งจีโนมมาตั้งแต่เริ่มการระบาดเมื่อ 3 ปีที่แล้วด้วยเหตุผลหลายประการ:
1. การติดตามสายพันธุ์ของไวรัส: โดยการถอดรหัสพันธุกรรมไวรัสทั้งจีโนมจากผู้ติดเชื้อหรือผู้ป่วยที่หลากหลาย จะช่วยให้นักวิทยาศาสตร์สามารถติดตามวิวัฒนาการของไวรัสและระบุสายพันธุ์ใหม่ได้ สายพันธุ์ย่อยที่อุบัติขึ้นมาอาจมีลักษณะที่แตกต่างกัน เช่น การแพร่เชื้อที่เพิ่มขึ้น หรือก่อความรุนแรงของการเจ็บป่วย และบางสายพันธุ์อาจหลีกเลี่ยงภูมิคุ้มกันที่สร้างขึ้นโดยการฉีดวัคซีนหรือการติดเชื้อตามธรรมชาติในครั้งก่อน
2. ทำความเข้าใจเกี่ยวกับการแพร่กระจายของไวรัส: การถอดรหัสพันธุกรรมไวรัสทั้งจีโนมสามารถให้ข้อมูลที่มีค่าเกี่ยวกับการแพร่กระจายของไวรัสภายในชุมชน ภูมิภาค และประเทศต่างๆ สามารถช่วยระบุกลุ่มผู้ป่วยและห่วงโซ่การแพร่เชื้อ ซึ่งสามารถใช้ปรับมาตรการด้านสาธารณสุขเข้าควบคุมการระบาดของไวรัสได้แบบเรียลไทม์
3. การป้องกันด้วยวัคซีนและการรักษาด้วยยา: หากไวรัสเกิดการเปลี่ยนแปลงที่สำคัญ (การกลายพันธุ์) สิ่งเหล่านี้อาจส่งผลกระทบต่อประสิทธิภาพของวัคซีนและการรักษาด้วยแอนติบอดีสำเร็จรูปและยาต้านไวรัส ทำให้นักวิจัยมีข้อมูลที่สามารถใช้อัปเกรดทั้งชุดตรวจ แอนติบอดีสำเร็จรูป วัคซีน และยาต้านไวรัส ได้ในระยะอันสั้นทันต่อเหตุการณ์
4. การตอบสนองด้านสาธารณสุข: ข้อมูลจากการถอดรหัสพันธุกรรมไวรัสทั้งจีโนมสามารถใช้ประกอบการตัดสินใจเกี่ยวกับการดำเนินการหรือยกเลิกมาตรการควบคุม ได้แบบเรียลไทม์ ตัวอย่างเช่น การถอดรหัสพันธุกรรมไวรัสทั้งจีโนม สามารถช่วยระบุได้ว่ามีการแพร่ระบาดของโควิด-19 สายพันธุ์ใหม่ที่สามารถแพร่เชื้อได้มากกว่าสายพันธุ์ดั้งเดิม ทางเจ้าหน้าที่อาจตัดสินใจเพิ่มความเข้มข้นในบางมาตรการ เช่น กินร้อน ช้อนกลาง สวมใส่หน้ากากอนามัย เว้นระยะห่างทางสังคม การใช้ยาแอนติบอดีสำเร็จรูปและยาต้านไวรัส รวมไปถึงการปิดพื้นที่ ตำบล จังหวัด หรือประเทศ
5. การทำงานร่วมกันทั่วโลก: การแบ่งปันข้อมูลจากการถอดรหัสพันธุกรรมไวรัสทั้งจีโนมทำให้นักวิทยาศาสตร์ทั่วโลกสามารถศึกษาไวรัส ติดตามการแพร่กระจาย การตอบสนองต่อภูมิคุ้มกันและยาต้านไวรัส รวมทั้งการเกิดเชื้อไวรัสดื้อยาได้อย่างมีประสิทธิภาพ ความร่วมมือระดับโลกนี้มีความสำคัญอย่างยิ่งต่อการควบคุมการแพร่ระบาด
6. การทำนายแนวโน้มในอนาคต: จากการสังเกตวิวัฒนาการของไวรัสเมื่อเวลาผ่านไป นักวิทยาศาสตร์สามารถคาดการณ์เกี่ยวกับแนวโน้มในอนาคต เช่น ความเป็นไปได้ที่สายพันธุ์ใหม่จะเกิดขึ้น สามารถหลบเลี่ยงภูมิคุ้มกันหรือเข้ายึดเกาะกับผิวเซลล์ได้ดีกว่าหรือด้อยกว่าสายพันธุ์ที่ระบาดในปัจจุบัน
โดยสรุปการถอดรหัสพันธุกรรมไวรัสทั้งจีโนมเป็นเครื่องมืออันทรงพลังที่ช่วยให้เราติดตามและทำความเข้าใจไวรัสโควิด-19 ได้แบบเรียลไทม์ เพื่อปรับเปลี่ยนการรับมือต่อการระบาดใหญ่ทั้งในระดับท้องถิ่นและระดับโลก ทั้งในปัจจุบันและอนาคต
- 378 views